
临床预测模型也是大家比较感兴趣的,今天就带着大家看一篇临床预测模型的文章,并且用一个例子给大家过一遍做法。
这篇文章来自护理领域*期刊的文章,文章名在下面
Ballesta-Castillejos A, Gómez-Salgado J, Rodríguez-Almagro J, Hernández-Martínez A. Development and validation of a predictive model of exclusive breastfeeding at hospital discharge: Retrospective cohort study. Int J Nurs Stud. 2021 May;117:103898. doi: 10.1016/j.ijnurstu.2021.103898. Epub 2021 Feb 7. PMID: 33636452.
文章作者做了个出院时单纯母乳喂养的预测模型,数据来自两个队列,一个队列做模型,另外一个用来验证。样本量的计算用的是“10个样本一个变量”的标准,预测结局变量是个二分类,变量筛选的方法依然是单因素有意义的都纳入预测模型中,具体的建模方法是logistic回归模型。然后用AUC进行模型的评估。
在结果报告上,作者报告了所有的有意义的预测因素,每个因素会展示OR和OR的置信区间,还有整体模型的评价指标,包括展示了模型的ROC曲线,和AUC,作者还展示了不同概率阶截断值下的模型的Sensitivity, Specificity, PPV, NPV, LR+, LR-。如图:
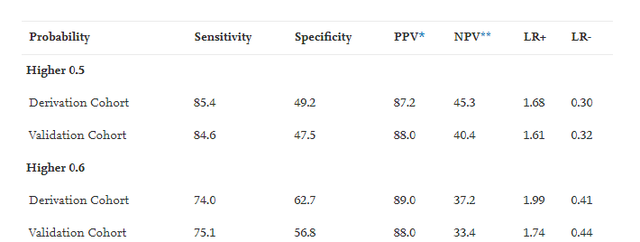
作者整个文章是用SPSS做出来的,今天给大家写写论文中的各种指标都是什么意义以及如何用R语言做出来论文中需要报告的各种指标。
理论铺垫
首先给大家写灵敏度(Sensitivity)与特异度(Specificity),这两个东西都是针对二分类结局来讲的,大家先瞅瞅下面的图:
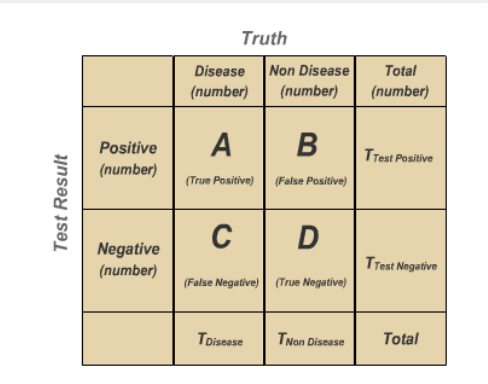
我们真实的结果有两种可能,模型的预测也有两种可能,上图的AD表示模型预测对的个案数量,那么灵敏度就是:在真的有病了,你的模型有多大可能检验出来,表示为
Sensitivity: A/(A+C) × 100
在论文中就是这个母亲真的是单纯母乳喂养的,模型有多大可能识别为真的单纯母乳喂养。
特异度就是:我是真的真的没病,你的模型有多大可能说我真的没病,表示为:
Specificity: D/(D+B) × 100
在论文中就是这个母亲真的不会去单纯母乳喂养的,模型有多大可能识别为真的不单纯母乳喂养。
有些同学说,我知道个模型预测准确率不就好了吗,用(A+D)/(A+B+C+D)来评估模型不就好了吗?搞这么麻烦。。
不能这么想的,比如你现在有一个傻瓜模型,这个模型傻到它只会将所有的人都预测为没病,刚好这个模型被用在了一个正常人群中,然后我们发现这个傻瓜模型的正确率也是100%,这个就很离谱,所以并模型预测准确性是不能全面评估模型表现的,需要借助Sensitivity, Specificity。
我们再看 PPV, NPV这两个指标:
Positive Predictive Value: A/(A+B) × 100
Negative Predictive Value: D/(D+C) × 100
看上面的公式,相信大家都看得出这两个其实就是模型的阳性预测准确性和阴性预测准确性,也可以从特定角度说明模型的表现。
再看LR+和 LR-,这两个就是阳性似然比 (positive likelihood ratio, LR+)和 阴性似然比
(positive likelihood ratio, LR+),似然比的概念请参考下一段英文描述:
Likelihood ratio (LR) is the ratio of two probabilities: (i) probability that a given test result may be expected in a diseased individual and (ii) probability that the same result will occur in a healthy subject.
那么:(LR+) = sensitivity / (1 - specificity),意思就是真阳性率与假阳性率之比。说明模型正确判断阳性的可能性是错误判断阳性可能性的倍数。比值越大,试验结果阳性时为真阳性的概率越大。
(LR-) = (1 - sensitivity) / specificity,意思就是假阴性率与真阴性率之比。表示错误判断阴性的可能性是正确判断阴性可能性的倍数。其比值越小,试验结果阴性时为真阴性的可能性越大。
所以大家记住:阳性似然比越大越好,阴性似然比越小越好。
最后再把上面的所有的内容总结一个表献给可爱的粉丝们,嘿嘿。下面就是一个分类结局预测变量需要报告的一些模型评估指标:
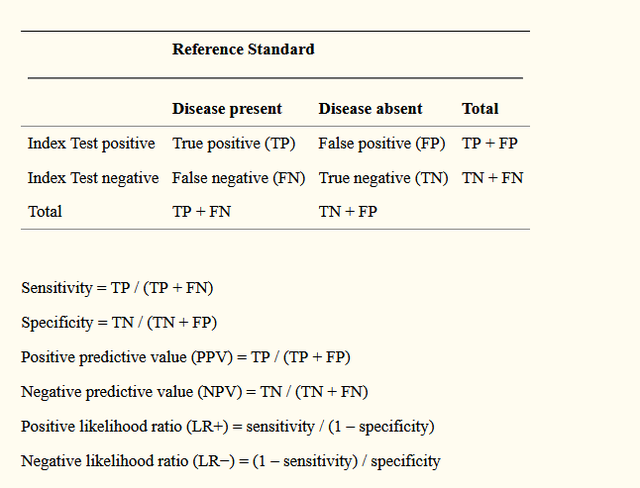
再回过头想想我们所谓的阳性或者阴性,如果用logistics回归做的话本身这个阳性阴性的判别都是可以设定的,因为我们的模型拟合出来的是响应概率,就是Logit公式里面的那个p值,你可以以p=0.5为我们判别阴阳性的cutoff,当然你还可以以0.9或者0.1为cutoff,cutoff不同自然我们模型灵敏度和特异度就不同了,就是说灵敏度和特异度是随着cutoff不同而变化着的,所以要稳定地评估模型表现还需要另外的指标,这个时候我们就引出了一个很重要的概念:ROC曲线和曲线下面积AUC。
在实战中理解ROC曲线
我现在手上有数据如下:
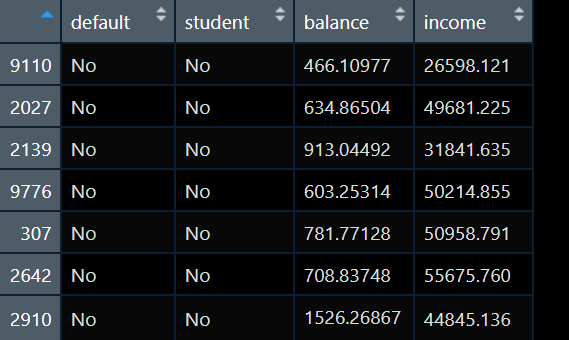
我要做一个default的预测模型,default是个二分类变量,取值为“No” 和“Yes”,为了简单我预测因素只考虑一个balance。于是我建立一个logistics模型:
model_glm = glm(default ~ balance, data = default_trn, family = "binomial")我们将预测为Yes的概率为0.1的时候作为cutoff值,划分预测结局(p<0.1的时候为No,p>0.1的时候为Yes),同样地我们还可以将cutoff设置为0.5,0.9。然后我们分别看一看模型的灵敏度和特异度:
test_pred_10 = get_logistic_pred(model_glm, data = default_tst, res = "default",
pos = "Yes", neg = "No", cut = 0.1)
test_pred_50 = get_logistic_pred(model_glm, data = default_tst, res = "default",
pos = "Yes", neg = "No", cut = 0.5)
test_pred_90 = get_logistic_pred(model_glm, data = default_tst, res = "default",
pos = "Yes", neg = "No", cut = 0.9)
test_tab_10 = table(predicted = test_pred_10, actual = default_tst$default)
test_tab_50 = table(predicted = test_pred_50, actual = default_tst$default)
test_tab_90 = table(predicted = test_pred_90, actual = default_tst$default)
test_con_mat_10 = confusionMatrix(test_tab_10, positive = "Yes")
test_con_mat_50 = confusionMatrix(test_tab_50, positive = "Yes")
test_con_mat_90 = confusionMatrix(test_tab_90, positive = "Yes")
metrics = rbind(
c(test_con_mat_10$overall["Accuracy"],
test_con_mat_10$byClass["Sensitivity"],
test_con_mat_10$byClass["Specificity"]),
c(test_con_mat_50$overall["Accuracy"],
test_con_mat_50$byClass["Sensitivity"],
test_con_mat_50$byClass["Specificity"]),
c(test_con_mat_90$overall["Accuracy"],
test_con_mat_90$byClass["Sensitivity"],
test_con_mat_90$byClass["Specificity"])
)
rownames(metrics) = c("c = 0.10", "c = 0.50", "c = 0.90")
metrics运行代码后得到响应概率不同cutoff值的情况下模型的灵敏度和特异度,如下图:
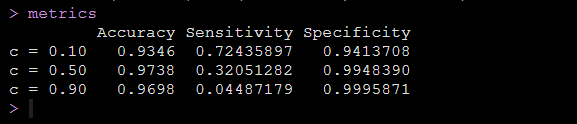
可以看到我们设定的响应概率的cutoff值不同,就是判断阴阳的标准不同,我们得到的模型的灵敏度和特异度就是不同的。
以上只是为了再次给大家直观地说明我们的模型的灵敏度和特异度是取决于我们的响应概率界值的,你判断阴阳的标准会直接影响模型的灵敏度和特异度,于是,我们换个想法,对于一个模型,我们将灵敏度作为横坐标,特异度作为纵坐标,然后cutoff随便取,我们形成一条曲线,这就考虑了所有的cutoff情况了,就可以稳定地评估模型的表现了,这条曲线就是ROC曲线。
那么我们期望的是一个模型它的灵敏度高的时候,特异度也能高,体现到曲线上就是曲线下的面积能够越大越好。
预测模型R语言实操
此部分给大家写如何做出论文中的各种指标,以及如何绘出ROC曲线。
依然是用上一部分的数据,依然是做balance预测default的模型:
model_glm = glm(default ~ balance, data = default_trn, family = "binomial")模型输出结果如下:
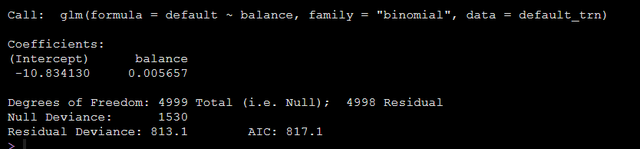
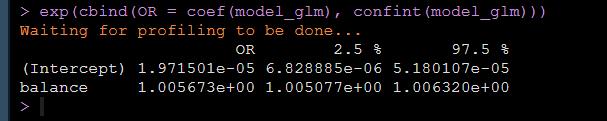
结果中有输出模型的截距和balance的β值,我们可以用如下代码得到balance的OR值以及置信区间:
exp(cbind(OR = coef(model_glm), confint(model_glm)))运行上面的代码就可以得到balance的OR值和OR的置信区间:
同时我们有原始的真实值,我们模型拟合好了之后可以用该模型进行预测,得到预测值,形成混淆矩阵:
model_glm_pred = ifelse(predict(model_glm, type = "response") > 0.5, "Yes", "No")在矩阵中就可以得到哪些是原始数据真实的No和Yes,哪些是模型预测出来的No和Yes:
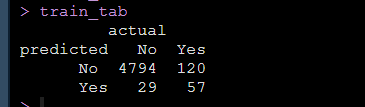
上面就是我们自己数据做出来的混淆矩阵,然后大家就可以直接带公式计算出需要报告的模型的Sensitivity, Specificity, PPV, NPV, LR+, LR-了。
同时大家可以用pROC包中的roc函数一行代码绘制出ROC曲线并得到曲线下面积,比如我做的模型,写出代码如下:
test_roc = roc(default_tst$default ~ test_prob, plot = TRUE, print.auc = TRUE)就可以得到ROC曲线和曲线下面积了:
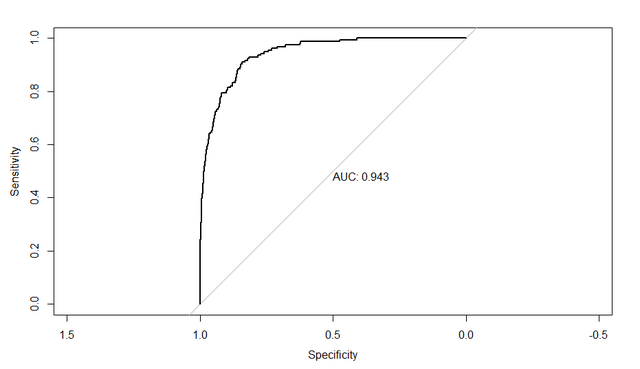
上面的所有操作都是可以在SPSS中完成的,论文作者也是用SPSS做的。大家感兴趣去阅读原论文哈。
小结
今天结合发表文章给大家写了分类结局的预测模型需要报告哪些指标,指标的意义以及如何用R做一个分类结局的预测模型,感谢大家耐心看完,自己的文章都写的很细,重要代码都在原文中,希望大家都可以自己做一做,请转发本文到朋友圈后私信回复“数据链接”获取所有数据和本人收集的学习资料。如果对您有用请先记得收藏,再点赞分享。
也欢迎大家的意见和建议,大家想了解什么统计方法都可以在文章下留言,说不定我看见了就会给你写教程哦,有疑问欢迎私信。R数据分析:跟随top期刊手把手教你做一个临床预测模型
